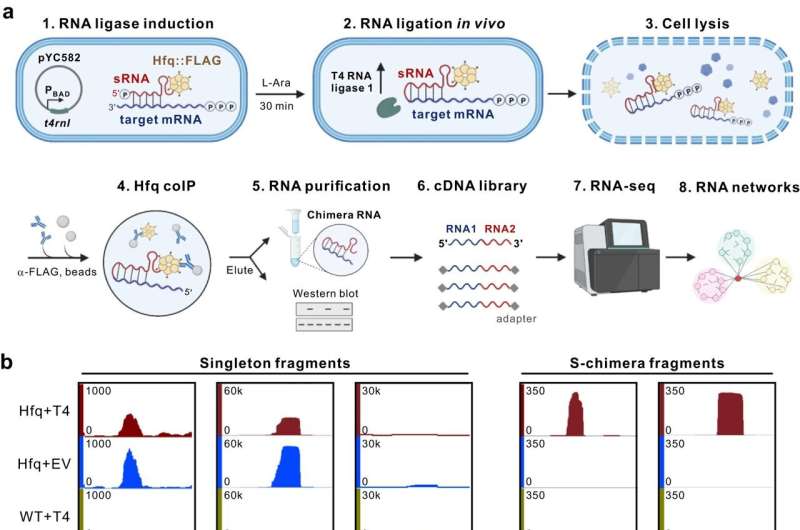
iRIL-seq captura fielmente las interacciones objetivo del ARNs. crédito: Comunicaciones de la naturaleza (2023). doi: 10.1038/s41467-023-43632-1
en un estudio publicado en Comunicaciones de la naturalezael grupo del profesor Zhao Yanjie en el Instituto de Inmunidad e Infecciones de Shanghai de la Academia de Ciencias de China informó sobre una nueva tecnología para la elaboración de perfiles de interacción de ARN (iRIL-seq), que identifica las interacciones moleculares ARN-ARN en células microbianas vivas.
Utilizando iRIL-seq, los investigadores han mapeado por primera vez las redes de interacción ARN-ARN de Salmonella enterica en diferentes etapas de desarrollo, descubriendo varios ARN no codificantes novedosos e importantes centros reguladores de ARNm.
La interacción directa ARN-ARN es el principal mecanismo a través del cual los ARN no codificantes ejercen sus funciones. En las células procarióticas, los ARN pequeños no codificantes reconocen los ARNm diana mediante interacciones de emparejamiento de bases muy cortas e incompletas, que son difíciles de predecir y cuantificar.
Los ARN bacterianos no codificantes tienen importantes funciones reguladoras, controlan la expresión de muchos genes diana en el nivel postranscripcional y desempeñan funciones críticas en muchos procesos biológicos, como la infección bacteriana, la resistencia a los antibióticos, el metabolismo central, la respuesta al estrés y la detección de quórum. ., y formación de biopelículas.
El análisis de la red de interacción ARN-ARN es de gran importancia para el estudio de la fisiología microbiana y las funciones del ARN no codificante. Sin embargo, las técnicas actuales de elaboración de perfiles de interacción ARN-ARN son complejas y requieren células fijas y numerosos pasos in vitro, como la digestión, la reparación y el empalme del ARN, lo que da como resultado interacciones de ARN insuficientes con baja especificidad y precisión.
En este estudio, los investigadores desarrollaron un nuevo enfoque de ligadura de proximidad de ARN in vivo, iRIL-seq (interacción intercelular de ARN mediante ligadura y secuenciación).
Los investigadores primero estimularon la expresión de la enzima ARN T4 en células microbianas vivas, lo que dio como resultado una unión de proximidad entre moléculas de ARN que interactúan in vivo. Luego enriquecieron pequeños ARN no codificantes y sus transcripciones que interactúan mediante inmunoprecipitación de la chaperona principal de ARN pequeño Hfq.
También determinaron la identidad y las secuencias de las moléculas de ARN que interactúan mediante secuenciación de ARN de alto rendimiento. El proceso experimental principal se puede completar en un día, gracias a una rápida reticulación in vivo y un flujo de trabajo simplificado.
Esta nueva tecnología no solo aclara el atlas de la red microbiana de interacción ARN-ARN, sino que también identifica las interacciones de emparejamiento de bases ARN-ARN con una resolución de un solo nucleótido. iRIL-seq es una técnica sencilla, rápida, precisa y universal para analizar las interacciones ARN-ARN en microorganismos y propone una nueva forma de estudiar las interacciones ARN-ARN en células eucariotas.
Además, los investigadores mapearon por primera vez las interacciones dinámicas ARN-ARN del patógeno bacteriano Salmonella enterica en múltiples etapas de desarrollo, identificaron más de 2000 interacciones ARN-ARN y descubrieron varios centros reguladores clave de ARNm. Se ha demostrado que el mayor centro regulador de ARNm en bacterias hasta la fecha es el ARNm que codifica la porina OmpD de la membrana externa, que está regulada por al menos 12 pequeños ARN no codificantes.
La caracterización experimental de estos pequeños ARN condujo a la identificación de un nuevo ARN pequeño FadZ escindido del ARNm 3’UTR por la ribonucleasa. La expresión del ARNm de FadZ y fadBA parental estaba regulada por los factores de transcripción aguas arriba FadR y CRP y la señalización de ácidos grasos de cadena larga.
En condiciones de ácidos grasos de cadena larga, tanto el ARNs de FadZ como el gen diana ompD fueron activados por el factor de transcripción CRP, formando juntos un bucle de alimentación incoherente de tipo I (I1-FFL). Los resultados revelaron el mecanismo regulador postranscripcional de las bacterias en respuesta al metabolismo de los ácidos grasos de cadena larga en los huéspedes.
más información:
Fang Liu et al., El perfil de interacción de ARN in vivo revela pequeños ARN procesados con 3’UTR que se dirigen a un centro regulador central, Comunicaciones de la naturaleza (2023). doi: 10.1038/s41467-023-43632-1
La frase: Una nueva técnica para analizar las interacciones ARN-ARN en células microbianas vivas (11 de diciembre de 2023) Obtenido el 11 de diciembre de 2023 de https://phys.org/news/2023-12-technique-rna-rna-interactions-microbian- células . lenguaje de programación
Este documento está sujeto a derechos de autor. Sin perjuicio de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin permiso por escrito. El contenido se proporciona únicamente con fines informativos.






More Stories
El jefe de la Agencia Espacial de EE.UU. quiere hablar con China sobre la basura espacial
Búsqueda de gemas: caracterización de seis planetas gigantes que orbitan enanas frías
La Administración Federal de Aviación de EE. UU. ha puesto en tierra los cohetes Falcon 9 de SpaceX en espera de una investigación sobre un raro accidente de aterrizaje frente a la costa.